结直肠癌是起源于结直肠上皮细胞的恶性肿瘤,其发病率和死亡率分别居于所有恶性肿瘤的第三位和第二位[1]。早期结直肠癌治疗后5年生存率可超过90%,甚至可以完全治愈,晚期患者的5年生存率不足10%[2],因此,结直肠癌的早诊早治是降低发病率和死亡率的关键,目前,多种类型的结直肠癌无创诊断标记物正被广泛研究。
结直肠包括盲肠、升结肠、横结肠、降结肠、乙状结肠和直肠部分。研究表明发生于不同部位的结直肠癌在流行病学、组织学类型、分子特征、疾病进展和预后等方面具有很大的差异,其原因可能是不同的发育起源或差异化的致癌因素(如细菌群落、营养供应和胆汁酸水平等的差异)[3,4]。本文将简要介绍基于解剖学位置的结直肠癌的分类、不同位置的结直肠癌的流行病学、癌变途径及相关分子特征的差异,最后强调在开发结直肠癌诊断标志物时应充分考虑位置差异。
在胚胎发育起源上,盲肠、阑尾、升结肠、肝曲、横结肠近端三分之二起源于中肠,而远端三分之一横结肠、脾曲、乙状结肠、降结肠结肠和直肠起源于后肠。左右结肠的分层理论早在1990年就由美国肿瘤专家Bufill等从分子遗传学角度提出[5],即根据肿瘤接近或远离结肠脾曲将结直肠癌分为近端肠癌或远端肠癌,之后的很多学者延用了这种划分方法,将结直肠癌分为两类:近端(右侧)结直肠癌为位于盲肠、升结肠和横结肠上的癌症;远端(左侧)结直肠癌为位于降结肠、乙状结肠和直肠上的癌症。之后也有研究者提出应该将直肠癌单独作为一个分类[6,7],因为其流行和预后不同于结肠癌,基于此,结直肠癌有三个位置分类,即右侧/近端结肠癌、左侧/远端结肠癌和直肠癌(图1)。
图1 结直肠癌的三个位置分类[7]
不同位置分类结直肠癌的流行病学、
癌变途径及分子特征
在流行病学方面,早发(发病年龄<50岁)结直肠癌和家族性腺瘤性息肉病(FAP)患者多发生在左侧/远端结肠和直肠;
在分子特征方面,右侧/近端结肠癌多富集高微卫星不稳定性(MSI-high)、高CpG岛甲基化表型(CIMP-high)和BRAF突变等特征,左侧/远端结肠癌多富集染色质不稳定(CIN)特征[7,8]。
如表1所示,结直肠癌的两种主要癌变途径分别为传统途径和锯齿状途径,其癌前病变类型分别为传统腺瘤和无蒂锯齿状病变(SSL)。传统腺瘤包括管状腺瘤、绒毛状腺瘤和管状绒毛状腺瘤,较为均匀地分布在左右结肠;而无蒂锯齿状病变则主要分布在右侧/近端结肠,由于其扁平或无柄的形态,且不易出血[9],无蒂锯齿状病变较传统腺瘤更难被肠镜或便隐血检测发现,无蒂锯齿状病变的漏检是间期癌发生的重要原因。
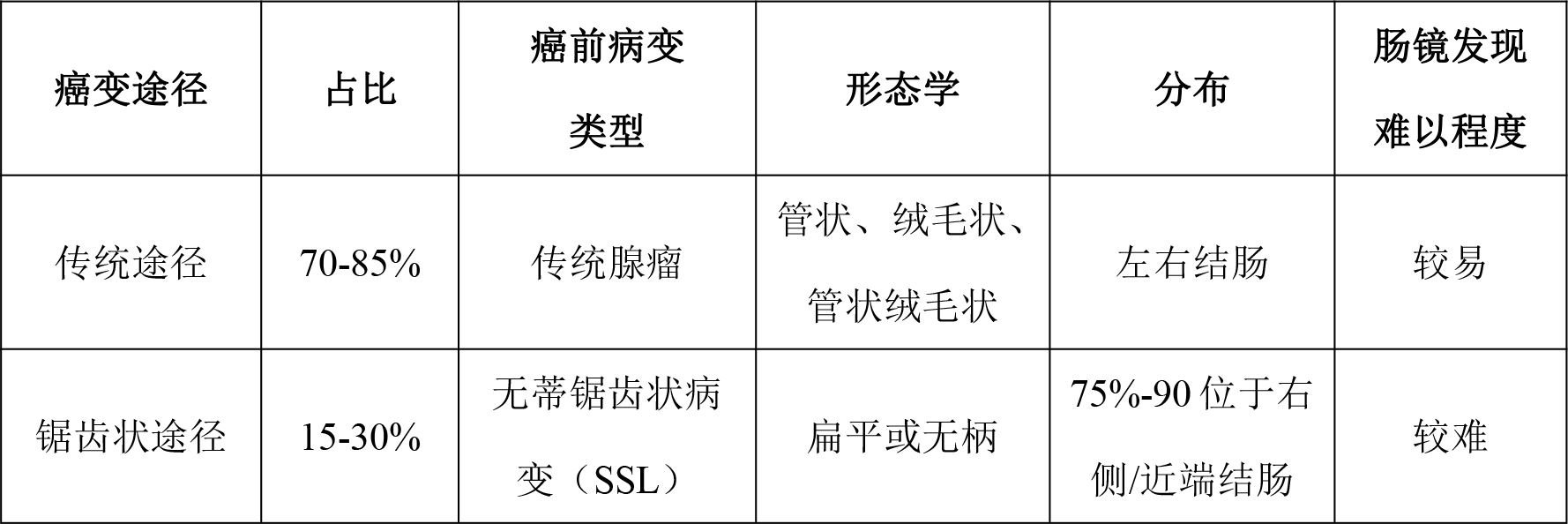
表1 结直肠癌传统和锯齿状癌变途径的差异
现有肠癌标记物的发现过程大多基于肠癌样本,无法考虑癌变途径,例如锯齿状形态仅保留在通过锯齿状路径产生的约三分之一的结直肠癌中,而其余三分之二在进展为晚期癌症时失去锯齿状组织学特征[10-13],这些问题使得难以通过形态学(即内镜和组织学)区分给定的结直肠癌是通过传统还是锯齿途径发展而来的,最终可能会导致筛选到的标记物的检测效果存在位置差异。
以结直肠癌常见的甲基化标记物SDC2和TFPI2为例,在一项研究中[14],研究者分析TCGA和GEO数据库中的结直肠癌样本及该研究收集到的临床组织样本,根据SDC2和TFPI2的甲基化水平,将样本分为HH组(SDC2和TFPI2均高甲基化)、HL组(一个基因高甲基化,另一个低甲基化)和LL组(SDC2和TFPI2均低甲基化),分析这几个组的样本在肿瘤位置方面的差异(图2)。结果显示HH组和LL组在右半结肠癌、左半结肠癌和直肠癌中均有分布,而HL组样本则主要分布在左半结肠癌样本中,其中HL中TFPI2高甲基化出现频率约是SDC2出现高甲基化频率的三倍,表明SDC2低甲基化主要发生在左半结肠癌中,而TFPI2在SDC2低甲基化样本中仍能保持高甲基化,进一步提示了SDC2对左半结肠癌的检出率可能不如右半结肠癌,而TFPI2的加入可能会弥补SDC2的漏检。
图2 HH、LL和HL三种基因型结直肠癌的位置分布
在另外两项研究中[15,16],研究者分别进行了多中心(图3和图4)的组织样本和/或粪便样本研究,结果均显示SDC2和TFPI2甲基化联合检测能提升对结直肠癌和腺瘤的检测敏感性,且敏感性的提升在左半结肠更为明显。
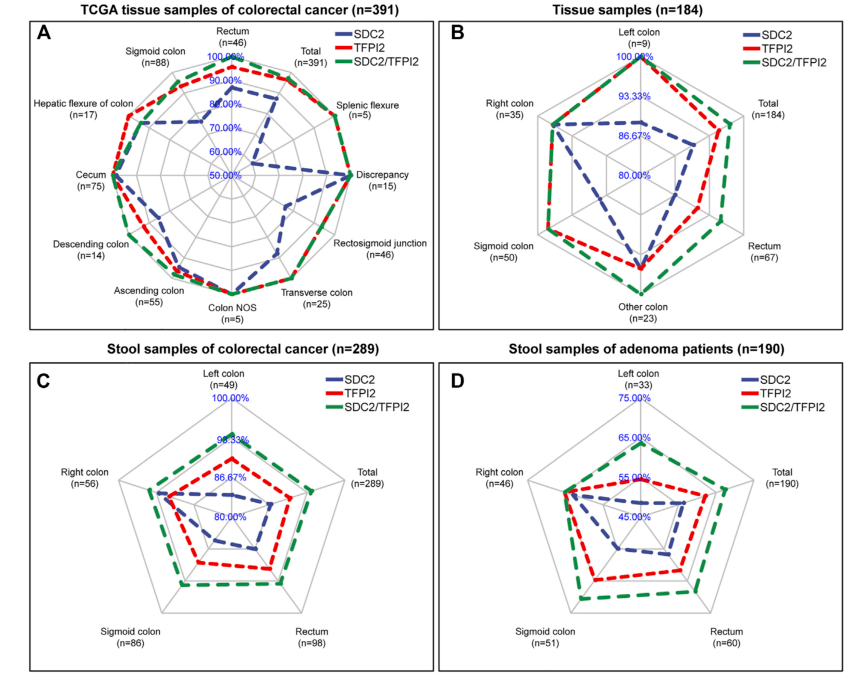
图3 SDC2和TFPI2联合检测互补效应与位置关系雷达图
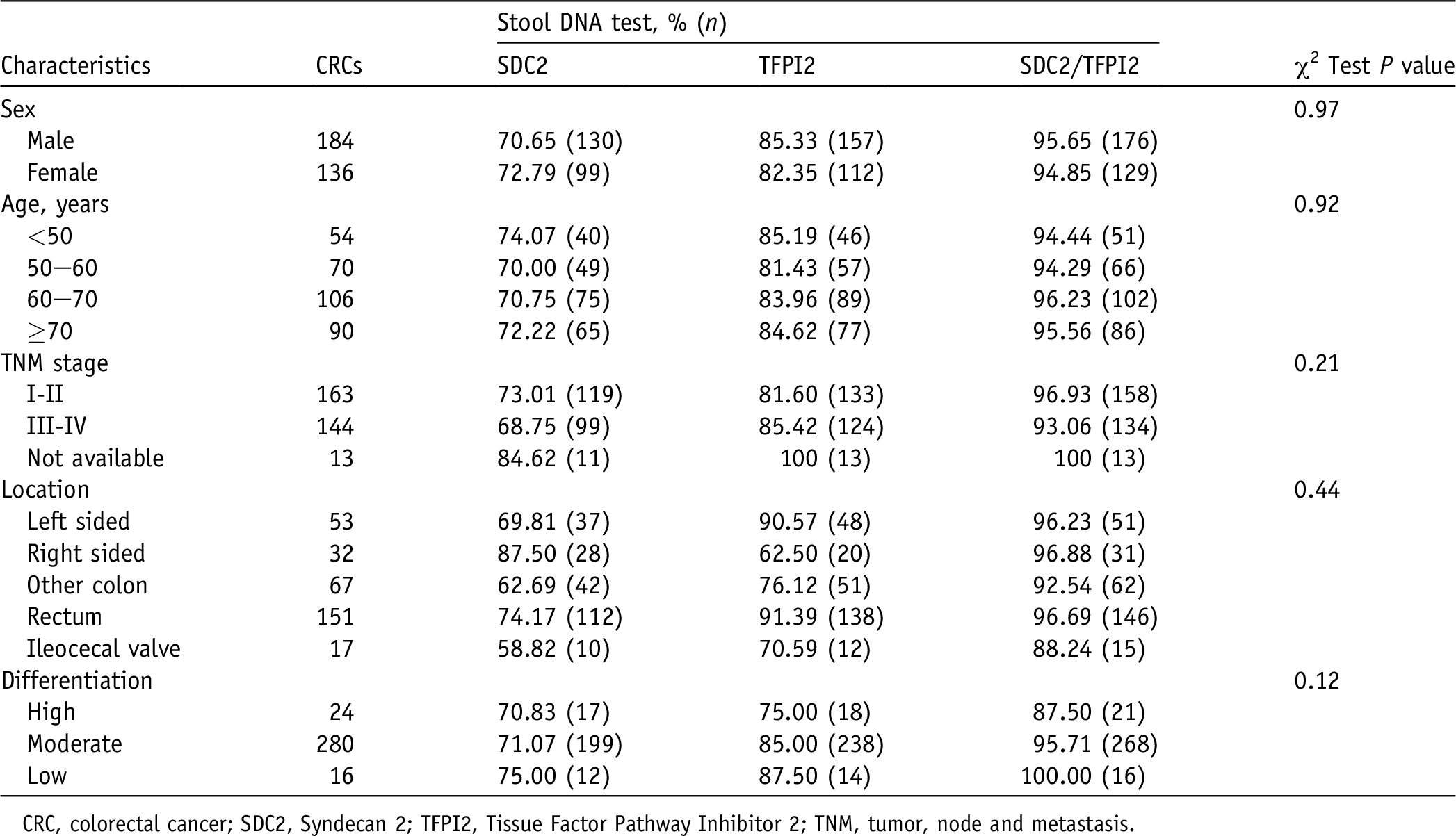
图4 SDC2和TFPI2甲基化在不同部位的结直肠癌中的检测敏感性分析
由前述可知,不同位置的结直肠癌对应着分子特征和癌变途径的差异,因此,在结直肠癌诊断标记物发现过程中应充分考虑这种位置差异,以便发现“一网打尽”式的全肠道结直肠癌检测标记物/标记物组合。
1.Sung H, Ferlay J, Siegel RL, et al. Global Cancer Statistics 2020: GLOBOCAN Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries. CA Cancer J Clin. 2021;71(3):209-249.
2.Siegel RL, Miller KD, Goding Sauer A, et al. Colorectal cancer statistics, 2020. CA Cancer J Clin. 2020;70(3):145-164.
3. Benedix F, Kube R, Meyer F, et al. Colon/Rectum Carcinomas (Primary Tumor) Study Group. Comparison of 17,641 patients with right- and left-sided colon cancer: differences in epidemiology, perioperative course, histology, and survival. Dis Colon Rectum. 2010;53(1):57-64.
4.Glebov OK, Rodriguez LM, Nakahara K, et al. Distinguishing right from left colon by the pattern of gene expression. Cancer Epidemiol Biomarkers Prev. 2003 ;12(8):755-62.
5.Bufill JA. Colorectal cancer: evidence for distinct genetic categories based on proximal or distal tumor location. Ann Intern Med. 1990; 113(10):779-88.
6.Li FY, Lai MD. Colorectal cancer, one entity or three. J Zhejiang Univ Sci B. 2009;10(3):219-29.
7.Keum N, Giovannucci E. Global burden of colorectal cancer: emerging trends, risk factors and prevention strategies. Nat Rev Gastroenterol Hepatol. 2019;16(12):713-732.
8.Baran B, Mert Ozupek N, Yerli Tetik N, et al. Difference Between Left-Sided and Right-Sided Colorectal Cancer: A Focused Review of Literature. Gastroenterology Res. 2018 ;11(4):264-273.
9. Benedix F, Kube R, Meyer F, et al. Colon/Rectum Carcinomas (Primary Tumor) Study Group. Comparison of 17,641 patients with right- and left-sided colon cancer: differences in epidemiology, perioperative course, histology, and survival. Dis Colon Rectum. 2010;53(1):57-64.
10.Bettington M, Walker N, Clouston A, et al. The serrated pathway to colorectal carcinoma: current concepts and challenges. Histopathology. 2013 Feb;62(3):367-86.
11.García-Solano J, Pérez-Guillermo M, Conesa-Zamora P, et al. Clinicopathologic study of 85 colorectal serrated adenocarcinomas: further insights into the full recognition of a new subset of colorectal carcinoma. Hum Pathol. 2010 Oct;41(10):1359-68.
12.Mäkinen MJ, George SM, Jernvall P, et al. Colorectal carcinoma associated with serrated adenoma--prevalence, histological features, and prognosis. J Pathol. 2001 Mar;193(3):286-94.
13.Tuppurainen K, Mäkinen JM, Junttila O, et al. Morphology and microsatellite instability in sporadic serrated and non-serrated colorectal cancer. J Pathol. 2005 Nov;207(3):285-94.
14.Lei R, Zhao Y, Huang K, et al. The methylation of SDC2 and TFPI2 defined three methylator phenotypes of colorectal cancer. BMC Gastroenterol. 2022;22(1):88.
15.Zhang L, Dong L, Lu C, et al. Methylation of SDC2/TFPI2 and Its Diagnostic Value in Colorectal Tumorous Lesions. Front Mol Biosci. 2021;8:706754.
16.Wang Z, Shang J, Zhang G, et al. Evaluating the Clinical Performance of a Dual-Target Stool DNA Test for Colorectal Cancer Detection. J Mol Diagn. 2022;24(2):131-143.